library(tidyverse)
library(arm)
library(readr)1 Introducción
Carga de paquetes:
Ejemplo de datos con medidas repetidas:
Conjunto de datos longitudinal de unos 2000 adolescentes australianos cuyos patrones de consumo de tabaco se registraron cada seis meses (a través de un cuestionario) durante un período de tres años.
Carga de datos:
smoking <- read_delim('./data/ARM_Data/smoking/smoke_pub.dat') Rows: 8730 Columns: 5
── Column specification ────────────────────────────────────────────────────────
Delimiter: "\t"
dbl (5): newid, sex(1=F), parsmk, wave, smkreg
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.smoking <- smoking %>% rename(sex = `sex(1=F)`) %>%
mutate(sex = factor(sex))Y graficamos la prevalencia en del fumado en cada una de las aplicaciones del cuestionario (wave) para cada uno de los sexos:
prevalencia <- smoking %>%
group_by(sex,wave) %>%
summarise(prevalencia = mean(smkreg)) `summarise()` has grouped output by 'sex'. You can override using the `.groups`
argument.ggplot()+geom_line(data=prevalencia,aes(x=wave,y=prevalencia,color = sex)
)+theme_minimal()+labs(title = 'Prevalencia de fumado por sexo',
x = 'Aplicación del cuestionario',
y = 'Prevalencia de fumado')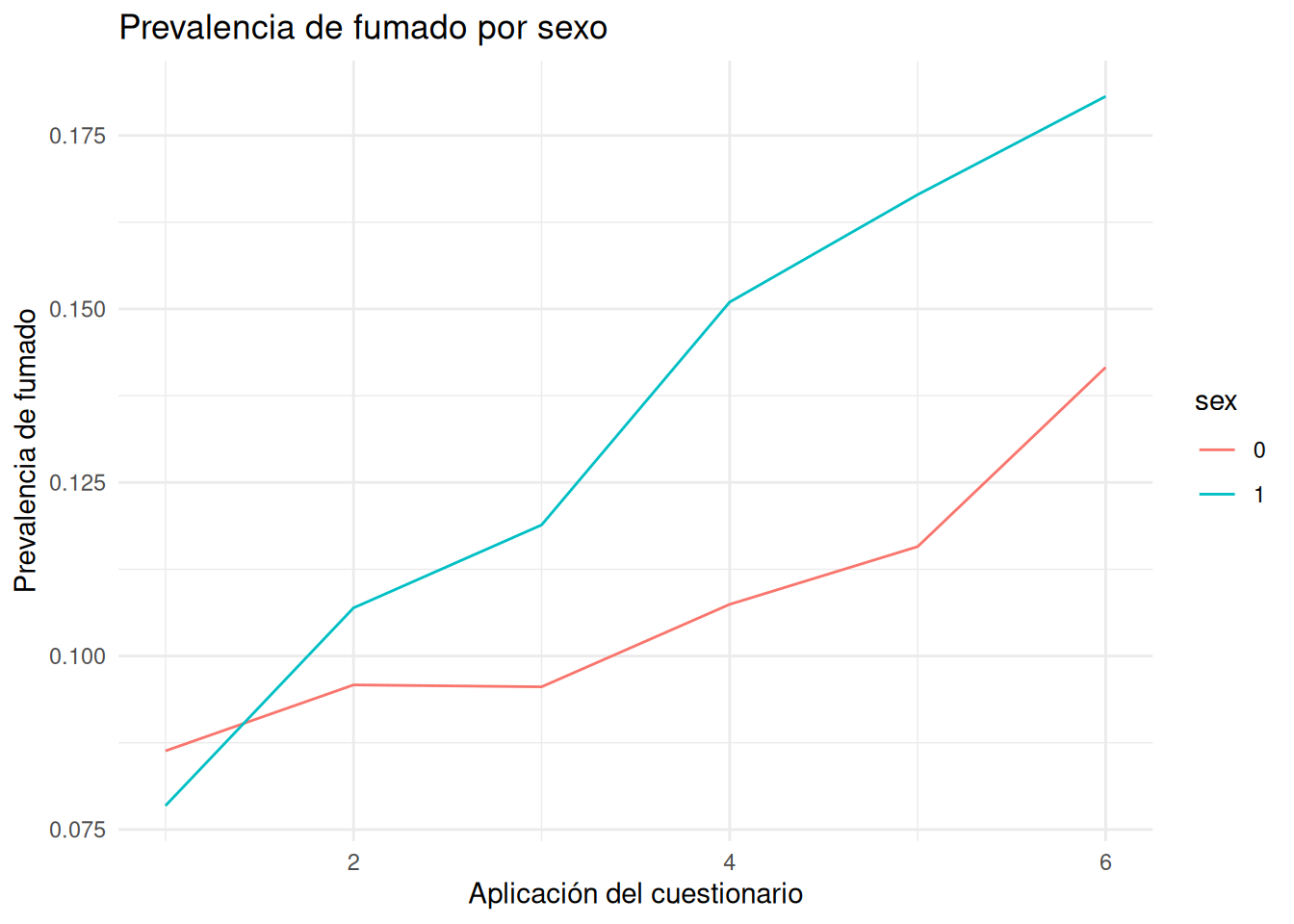
También podemos separar la información propia del nivel 1 (waves) y los de nivel más agrupado o nivel 2 (individuos):
smoking_ind <- smoking %>%
dplyr::select(wave,newid,smkreg) %>%
arrange(wave,newid)
head(smoking_ind)# A tibble: 6 × 3
wave newid smkreg
<dbl> <dbl> <dbl>
1 1 1 0
2 1 2 0
3 1 3 0
4 1 4 0
5 1 5 0
6 1 6 0smoking_group <- smoking %>%
dplyr::select(newid,sex,parsmk) %>%
distinct()
head(smoking_group)# A tibble: 6 × 3
newid sex parsmk
<dbl> <fct> <dbl>
1 1 1 0
2 2 0 0
3 3 1 0
4 4 1 0
5 5 0 0
6 6 0 0